背根神经节(dorsal root ganglia,DRG)作为感觉神经元胞体的聚集地,承担着将感觉信号从外周传入中枢的重要功能。神经嵴细胞(neural crest cell, NCC)可以分化为DRG中的多种感觉神经元和神经胶质细胞,这一过程需要特异的信号分子和转录因子的精细调控。目前,对DRG发育和细胞类型及功能的研究集中在小鼠模型上,然而,人类感觉神经元在亚型和功能基因表达上与小鼠存在一定的差异,因此,对于人类DRG发育的特点和细胞命运决定的调控机制还有待于进一步研究。此外,类器官培养近年来受到广泛关注,它可以通过体外对干细胞的培养和有序分化,实现与相应的器官类似的空间组织,并能够重现相应器官的部分功能,为研究人员提供了研究器官发育和疾病的有效平台。因此,通过深入解析人DRG发育的调控机制,并在体外建立DRG类器官模型将为人类感觉神经元发育和生理功能的探究提供理想工具。
2024年11月12日,北京师范大学吴倩课题组与广东省智能科学与技术研究院张旭院士课题组, 新基石研究员王晓群课题组合作,在Cell杂志上发表了题为Decoding transcriptional identity in developing human sensory neurons and organoid modeling的研究论文。研究团队深入解析了人类背根神经节发育过程中调控多种感觉神经元分化的多层级信号通路,建立人类DRG类器官模型,并利用该模型对调控感觉神经元谱系发育的转录因子进行验证。此外,该研究还发现了一种人类特有的伤害感受器细胞亚型,并在人DRG类器官中复现了该类细胞的发育和功能(图1)。
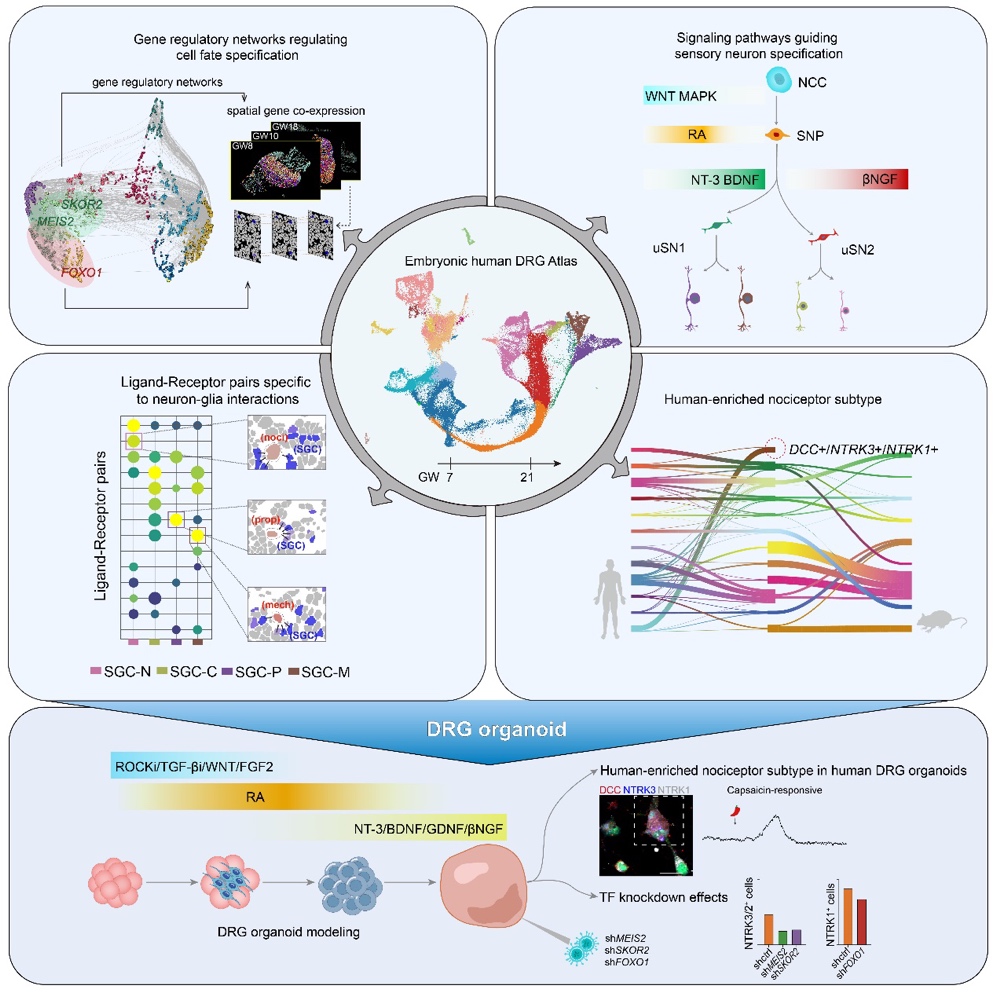
图1.人类背根神经节时空发育机制和类器官模型构建
为了构建人类DRG类器官模型,研究团队使用基于单分子成像的单细胞空间转录组技术TF-seqFISH(Transcription Factor sequential Fluorescence In Situ Hybridization)(1,2),并结合单细胞转录组测序技术,对妊娠早中期人类胚胎DRG发育进行全面解析。研究发现NCC经历两个神经发生的关键时间,并前后产生两种仍未特定分化的感觉神经元(unspecialized sensory neuron, uSN), uSN1和uSN2, 这些未特定分化的神经元未来会进一步受到转录因子的调控,分别分化为大直径的感觉细胞和小直径的感觉细胞,是感觉神经元多样性的基础。通过解析早期NCC谱系分化轨迹,研究人员深入阐明了参与uSN命运决定过程的外部信号通路和内部转录因子调控的联合机制。
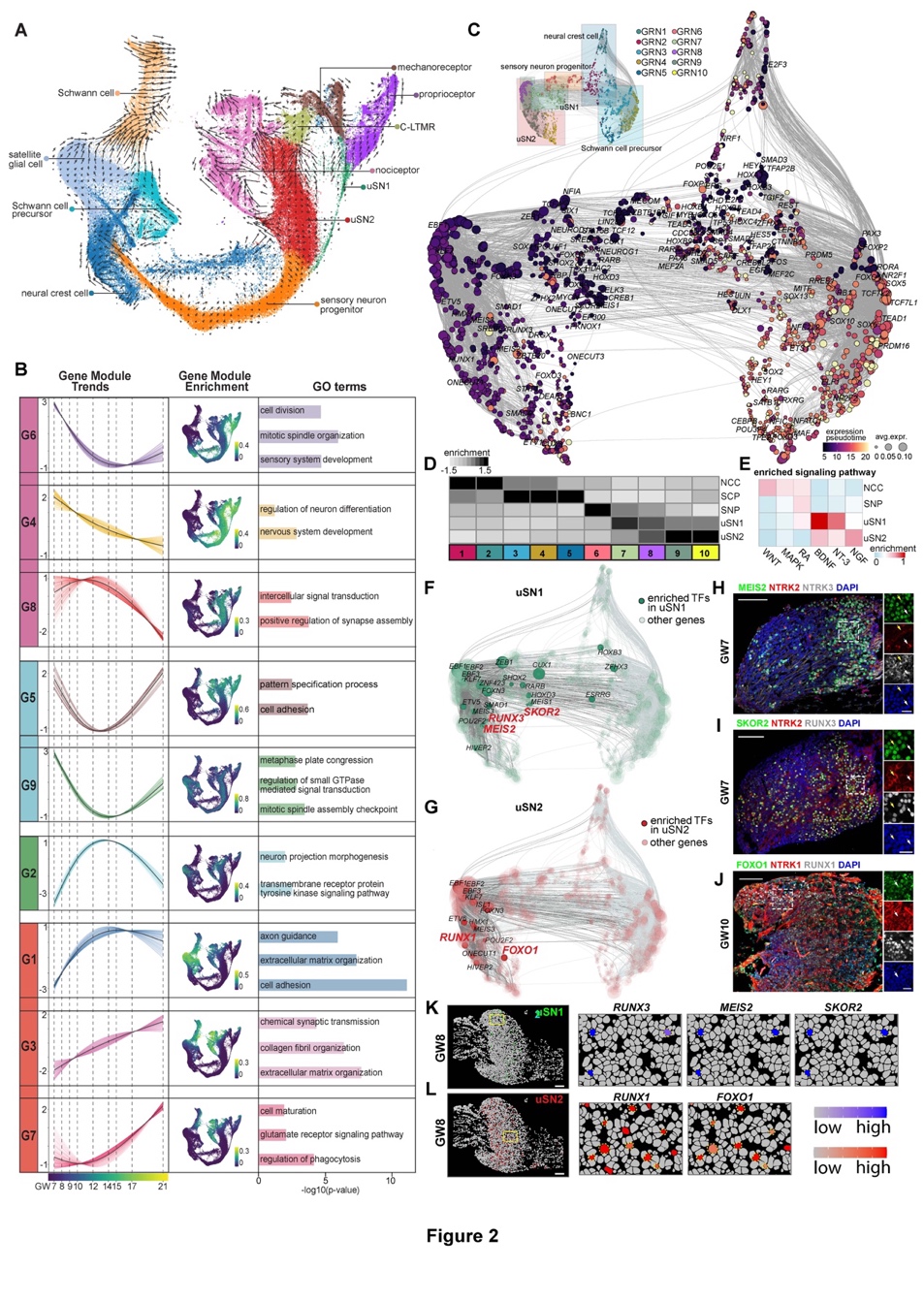
图2.神经元分化的信号调控机制
基于以上多层级信号通路在DRG神经元命运决定中的重要作用,研究人员通过时序性的加入不同的信号因子,成功的构建了功能性背根神经节类器官(hDRGOs),在体外复现了多能干细胞-NCC-感觉神经祖细胞-感觉神经元的分化过程,其中包括人类背根神经节中主要三类感觉神经元。这些神经元从发育轨迹到基因表达,都与人DRG的发育非常相似。此外,这些感觉神经元呈现假单极形态,并对辣椒素的刺激有相应的生理响应,进一步证明该DRG类器官可以获得具有一定功能的感觉神经元。
通过物种间比较,研究人员发现人类和小鼠在感觉神经元的发育进程、基因表达谱和细胞亚型上均存在差异。研究发现一类在人类DRG中特异富集的伤害感受器亚型,以DCC/NTRK3/NTRK1基因的表达为特征,这一群体在发育和成年的人DRG中均特异性存在。此外,研究人员发现hDRGOs中也存在该类感觉神经元亚型,进一步联合钙成像,证明了这类感觉神经元可以被辣椒素特异激活,通过体外实验明确了这类细胞是伤害感受器的一种亚型。此外,研究人员还利用DRG类器官模型验证了多种转录因子在调控感觉神经元分化谱系中的重要作用,表明类器官在人类感觉神经元发育和功能研究中具有重要的价值。
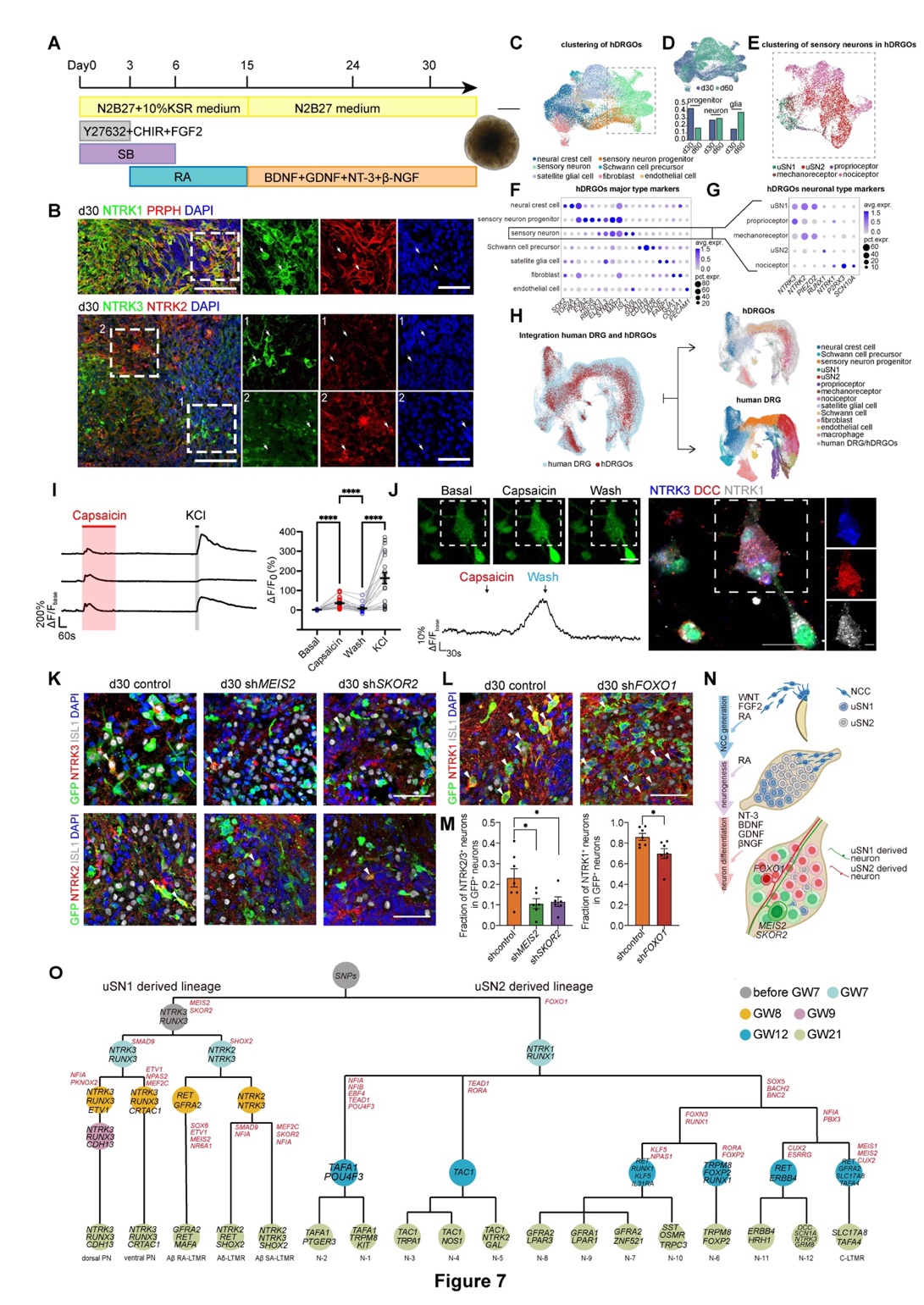
图3. 人背根神经节类器官
总之,该研究基于对人类DRG发育过程的解析,成功建立了人类背根神经节类器官模型,在体外模拟感觉神经元多样性和功能建立过程,并应用类器官探究了人类感觉神经元发育调控和人性进化上的特征。本研究为人类胚胎背根神经节研究提供了重要的时空转录组数据,建立的人类背根神经节类器官模型对感觉神经元发育和相关疾病的研究具有重要的意义。
新基石研究员王晓群教授、北京师范大学吴倩教授和广东省智能科学与技术研究院张旭研究员为该论文的共同通讯作者。中科院生物物理研究所博士研究生卢甜和博士研究生王梦迪为该论文的共同第一作者。
论文链接:https://doi.org/10.1016/j.cell.2024.10.023
参考文献:
1. Shi Y, Huang L, Dong H, Yang M, Ding W, Zhou X, Lu T, Liu Z, Zhou X, Wang M, Zeng B, Sun Y, Zhong S, Wang B, Wang W, Yin C, Wang X, Wu Q. Decoding the spatiotemporal regulation of transcription factors during human spinal cord development. Cell Res. 2024 Mar;34(3):193-213. doi: 10.1038/s41422-023-00897-x.
2. Zhong S, Wang M, Huang L, Chen Y, Ge Y, Zhang J, Shi Y, Dong H, Zhou X, Wang B, Lu T, Jing X, Lu Y, Zhang J, Wang X, Wu Q. Single-cell epigenomics and spatiotemporal transcriptomics reveal human cerebellar development. Nat Commun. 2023 Nov 22;14(1):7613. doi: 10.1038/s41467-023-43568-6.


